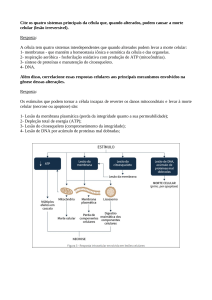
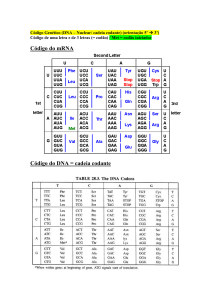
Aula de Bioquímica II Tema: Controle da Expressão Gênica Prof. Dr. Júlio César Borges Depto. de Química e Física Molecular – DQFM Instituto de Química de São Carlos – IQSC Universidade de São Paulo – USP E-mail: borgesjc@iqsc.usp.br Controle da Expressão Gênica Todas as células de um contêm as mesmas informações depositadas no genoma O Gene é expresso quando ele é transcrito em mRNA Principal ponto de controle Circuitos regulatórios guiam o desenvolvimento de eucariotos multicelulares e envolvem diversos tipos de mecanismos regulatórios A expressão de uma proteína pode ser: Constitutiva: Constantemente expressa Induzida: Expressa quando necessária Controle da Expressão Gênica Processos que afetam a concentração do produto gênico: pontos potenciais de controle Gene: É um segmento de DNA que leva à produção de um transcrito de RNA e inclui regiões que antecedem e que seguem a região transcrita, bem como sequências que não são traduzidas (íntrons) que se intercalam aos segmentos codificadores individuais (éxons), que são traduzidos. Inclui elementos reguladores, o promotor e oterminador da transcrição A iniciação da transcrição (1) é o principal mecanismo, pelo menos o mais bem documentado regulação sincronizada O processamento pós-transcricional (2) e a estabilidade do mRNA (3) são muito importantes, principalmente em eucariotos Controle da Expressão Gênica Em procariotos A identidade da sequência consenso do promotor dita a “força” da transcrição pela frequência do recrutamento da RNApol Existe modulação adicional por proteínas Esquema geral simplificado do promotor de genes de eucariotos Existem elementos regulatórios a milhares de bases a upstream do promotor Região promotora do gene da PEPcarboxiquinase Alta complexidade da regulação deste gene. Ajuste fino proporcional a importância do processo Controle da Expressão Gênica As informações para o controle da expressão gênica estão no DNA Regulação negativa Dependente de Repressores - Inibem a expressão gênica por bloquear o acesso da RNApol ao promotor - Dependem da presença do Efetor (sinal molecular) Tipo característico em procariotos Elementos operador próximos ao promotor Controle da Expressão Gênica As informações para o controle da expressão gênica estão no DNA Regulação positiva Dependente de Ativadores Induz a expressão Gênica - Dependem da presença do Efetor (sinal molecular) Tipo característico em eucariotos A Expressão Gênica é regulada por proteínas Proteínas específicas reconhecem sequências específicas de DNA Possuem domínios de interação com DNA e de interação com outras proteínas - Normalmente dímeros reconhecer sequências palindrômicas simétricas o papel dos Sulcos maior (mais específico) e menor (menos específico) no DNA Alta especificidade KA das proteínas regulatórias pelas sequências-alvo é 4-6 ordens de grandeza maior do que para outras sequências - Sequências específicas de DNA superfície específica para formação de ligações de H Domínios de Ligação ao DNA -Formam estruturas autônomas separadas do restante da proteína - Estruturas capazes de interagir estreita e especificamente com o DNA-alvo - Relativamente pequenos (60-90 resíduos) - projetam-se na superfície das proteínas - Conectadas ao restante da proteína por regiões flexíveis O Domínio Hélice-volta-hélice está presente em muitas proteínas ligadores de DNA - Apresentam-se como “dímeros” de forma a reconhecer a sequência palindrômica no DNA - Forma um eixo de simetria com DNA Domínios de Ligação ao DNA Organização dimérica (exceto o zinc finger) Reconhecimento por uma fita beta Zíper de Leucina Leu Hélice-alça-hélice Resíduos básicos Interação com o Pi Zinc Fingers Homeodomínios Controle da expressão em Procariotos Procariotos produzem proteínas relacionadas a processos interdependentes de forma agrupada - Mecanismo geral simples para coordenar a regulação de genes inter-relacionados Sistema óperon: unidade básica de transcrição - Operador sequência de DNA próximo ao promotor mRNA policistrônico: 1 promotor 1 mRNA várias proteínas traduzidas - Cada sequência codificadora tem seu próprio motivo Shine-Dalguarno - Existem exemplos deste sistema em eucariotos exceção Controle da expressão em Procariotos Procariotos induzem a expressão de enzimas conforme a disponibilidade de fonte de energia β-galactosidade é uma importante ferramenta biotecnológica atua sobre galactosídios alternativos Controle da expressão em procariotos A indução da expressão da β-galactosidase induz a expressão de 2 outras enzimas - Galactosídeo permease: transporte de lactose pela membrana - Tiogalactosídeo transacetilase: desintoxicação de outras moléculas transportadas pela permease Indução conjunta de enzimas para adaptação a um novo ambiente: Mecanismo comum de indução. Modelo do óperon: Explica a regulação paralela de diferentes proteínas -Formado por: - um gene regulador: codifica uma proteína repressora - uma sequência operadora: alvo da proteína repressora - genes estruturais: produtos da expressão do óperon operon Lac Conjunto de genes envolvidos na regulação e expressão das proteínas envolvidas no metabolismo de Lactose. P: sequência promotor lacI: gene proteína repressora O: operadores: sítio de ligação da proteína repressora lacZ: gene da β-galactosidade lacY: gene da permease lacA: gene da tio-transacetilase lacZ, lacY e lacA formam um mRNA policistrônico ou poligênico Proteína repressora Lac Permanece fortemente ligado à sequência operadora na ausência de Lactose - KA 1x106 vezes maior pela sequência operadora do que por outra sequência Impede a transcrição por impedir que a RNApol desenrole o DNA localmente Estrutura tetramérica Cada monômero possui um domínio hélice-voltahélice que reconhece a sequência operadora - Ocasiona a formação do loop de DNA Proteína repressora Lac O ligante da Proteína repressora Lac é um produto secundário da β-galactosidase 1,6-alolactose: INDUTOR - A lactose não interage com a Proteína repressora Lac α-D-galactopiranosil-(1 4)-D-glicopiranose Indutores α-D-galactopiranosil-(1 6)-D-glicopiranose Óperon Lac 1) Na ausência de Lactose, não existe 1,6-alolactose e portanto a Proteína repressora Lac permanece ligada à sequência operadora impedindo a transcrição do DNA 2) Na presença de Lactose, parte desta é convertida a 1,6-alolactose que se liga à Proteína Repressora Lac e reduz a afinidade desta pela sequência operadora. Com a saída da Proteína repressora Lac da sequência operadora os genes estruturais podem ser transcritos na forma de um mRNA policistrônico ou poligênico Mesmo na ausência de lactose, existe transcrição basal dos genes estruturais lacI mRNA Repressor lacI ligado ao operador bloqueia a transcrição dos genes lacZ, lacY e lacA lacI mRNA lac mRNA (policistrônico) Permease β-galactosidase Transacetilase Controle da Expressão Gênica em procariotos Comutação de reguladores Ativação da expressão gênica por AMPc AMPc (coativador) indução conjunta de enzimas catabólicas [Glicose] alta [AMPc] baixa e lactose ausente [Glicose] baixa [AMPc] alto e lactose ausente AMPc liga-se à cAMP receptor Protein – CRP CRP liga-se ao operador do operon Lac a -51 do Promotor e “recruta” a RNApol Atuam em vários óperons Regulons: rede de óperons com reguladores comuns [Glicose] alta [AMPc] baixo e lactose presente Ativação fraca [Glicose] baixa [AMPc] alto e lactose presente Ativação forte Efeito coordenado entre repressor/ativador Controle da Expressão Gênica em procariotos Em procariotos, muitos óperons funcionam de forma análoga ao óperon Lac Ex: óperon Pur Controla o metabolismo de Purinas A proteína Repressor Pur liga-se à sequência operadora na presença de um ligante: co-repressor O genoma de E. coli contêm mais de 20 sequências operadora para a proteína repressora Pur Regulon Permite a regulação paralela de todos os passos metabólicos da síntese de purinas Controle da Expressão Gênica em procariotos A estratégia dos atenuadores - Comum nos óperons para síntese aminoácidos Atenuação do óperon trp - Ocorre na síntese do mRNA O óperon trp controla a síntese de - Formação de grampos de mRNA auto complementares Triptofano Altos níveis de Trp reprime o óperon - Ligação do Trp a proteína repressora induz ligação ao operador Se a [Trp] ↑ síntese rápido do peptídeo líder Formação alça 3-4 fim da transcrição Se a [Trp] ↓ síntese lenta do peptídeo líder Formação alça 2-3 transcrição continua Controle da Expressão Gênica em procariotos Outros mecanismos, inclui-se a autoindução em biofilmes: quorum sensing Feedback de tradução de óperons de proteínas ribossomais - Visa regular a concentração das proteínas ribossomais e rRNA A proteína ribossomal inibe a tradução de seu próprio mRNA A medida que o respectivo rRNA é sintetizado, a proteína ribossomal dissocia-se de seu mRNA e liga ao rRNA - Maior afinidade pelo rRNA Controle da tradução Resposta estringente - Depende da disponibilidade de aminoácidos - Ausência de aminoácidos - Recrutamento do fator estringente - Síntese de ppGpp - Inibição da RNApol Controle da Expressão Gênica em procariotos Papel de pequenos RNAs sRNA Alguns mRNAs bacterianos são regulados por sRNA em cis ou em trans trans: outro sRNA regula a tradução de um dado mRNA Indução Função dependente da proteína Hfq facilita formação de duplex RNA-RNA cis: parte do próprio mRNA regula a própria tradução - Riboswitches: Aptâmeros naturais de RNA dependentes de ligantes - Ligantes identificados: TPP, cobalamina, FMN, Lys, Gly, purinas e adoMet (Sadenosilmetionina). Repressão Mecanismo de splicing em eucariotos Controle da Expressão Gênica em procariotos Controle por recombinação Recombinação regula a expressão de diferentes proteínas do flagelo (FljB e FliC) a cada 1000 gerações Mecanismo de escape do sistema imune Sistema óperon - Recombinase Hin inverte posição do promotor (e gene para a hin) para a FljB - Sem expressão da proteína repressora FljA expressão da FliC Controle da Expressão Gênica em Eucariotos O tamanho do genoma Diversidade Celular - Impõem mais complexidade para atingir especificidade. Transcrição e tradução NÃO são processos acoplados. Os genes de uma mesma via não estão organizados em óperons. - Existem somente algumas exceções Transcrição basal não observada transcrição está inativa para a maioria dos genes - Regulação positiva - Regulação negativa requer Ativadores sempre necessitaria de grande número de repressores presentes na célula em quantidade há poucos exemplos em eucariotos Papeis dos ativadores ou fatores de transcrição 1) Montagem do complexo de transcrição basal com os TFII 2) Outras proteínas regulatórias específicas se unem a este complexo basal - Ligam-se à sequências “Enhancer” - Potenciadores São proteínas modulares e multiméricas – Domínio de Ligação ao DNA e Domínio Ativador Remodelamento da Cromatina O DNA de eucariotos se apresenta ligado à proteínas formando os Nucleossomos - As regiões do DNA que estão sendo transcritas estão menos compactadas - Heterocromatina fortemente condensada: não disponível para transcrição - Eucromatina Parcialmente disponível para a transcrição A regulação negativa em eucariotos seria redundante com os nucleossomos Remodelamento da Cromatina A acetilação das histonas contribuem para a ativação da transcrição - Histonas acetilases (HAT) acetilam a Lys da cauda N-Terminal das Histonas 1) Reduz a afinidade da Histona pelo DNA afrouxa o nucleossoma 2) Recrutamento de outros componentes da maquinaria de transcrição 3) Iniciação da remodelagem da cromatina Reversão executada por Histonas desacetilases repressão da transcrição por compactação da cromatina Remodelamento da Cromatina Em levedura, os genes necessários para o metabolismo de galactose são ativados pela proteína GAL4. - Reconhece a sequência: 5’-CGG(N)11CCG-3’ 4.000 pontos no DNA de levedura - Somente 10 deles são identificados pela GAL4 99,75% estão bloqueados Regiões do DNA que estão mais ou menos compactadas são tecidos específicas Existem proteínas remodeladoras do DNA tecido específicas que regulam os conjuntos de genes que deverão ser transcritos e os que devem ser “silenciados”. O DNA é metilado por Metilases específicas no C5 da C - 70% das 5’-CG-3’ estão metiladas - A metila projeta-se para o sulco maior bloqueando interações - As 5’-CG-3’ próximas ao 5’ dos genes ativos são hipometiladas Controle da Expressão Gênica em Eucariotos As informações para o controle da expressão gênica estão no DNA Envolve interação, direta ou indireta, de ativadores/repressores e a RNApol em eucariotos Requerimento de: - elementos potenciadores (enhancers) a longa distância do promotor - Regulador de arquitetura para dobrar o DNA (alças ou looping) - Coativador aumenta a taxa de recrutamento/ativação da RNApol Repressor pode atuar bloqueando a ação do coativador Controle combinatório - Depende do subconjunto de proteínas produzidas naquele tipo celular - Heteroligômeros e heterocomplexos Controle da Expressão Gênica em Eucariotos Controle combinatório Dependendo do conjunto de proteínas diferentes efeitos Número reduzido de proteínas reguladoras maior especificidade reconhecem sequências assimétricas Ex: 2 famílias de reguladores com 3 membros cada Formam Homodímeros e heterodímeros 36 possíveis combinações Acentuadores ou potenciadores - sequências específicas “acima” do promotor que funcionam como pontos de ligação específicos para proteínas ativadoras ou repressoras. - Podem estar a milhares de pares de base à 5’ do promotor - Dependem da presença das proteínas ligadoras específicas - Podem ajudar a expor o DNA para RNApol II - Podem ajudar a montagem do complexo basal de transcrição Acentuador da creatina quinase regulando a expressão da β-galactosidade Regulação gênica em eucariotos Requer ação combinada de diversos agentes UAS: upstream activator sequences: Sequências ativadoras a montante Pode ser a jusante também Reguladores da arquitetura: proteínas HMG Remodelamento da cromatina Coativadores: Ativadores ou repressores interação com o Mediador Fatores basais de transcrição Regulação gênica positiva em eucariotos Coreografia da ativação da transcrição Eventos em cascata variável para diferentes genes Processo dependente de moléculas efetoras e reversível Expressão gênica regulada por hormônios Receptores Nucleares Os receptores para hormônios estereoidais atuam induzindo a transcrição - Proteínas modulares domínio de ligação ao DNA e domínio de ligação ao hormônio Ex: Estrogênio e testosterona ~ 50 membros no genoma humano Atuam como dímeros reconhecem elementos simétricos Agonistas Antagonistas Expressão gênica regulada por hormônios Receptores Nucleares Os Receptores de hormônios nucleares regulam a transcrição por recrutar Co-ativatores ou mediadores para o complexo de transcrição Expressão gênica regulada por hormônios Ação hormônios esteroides, tireoidianos e retinoide - Livre transito pela membrana plasmática Presença de sequências HRE: elementos de resposta hormonal específicos Regulação por repressão da Tradução Transcrição e tradução são processos desacoplados citoplasma edição do mRNA e transporte para o Envolve o armazenamento de mRNAs inativos no citoplasma 1) Fatores de iniciação da tradução são sujeitos a fosforilação inibe 2) Proteínas repressoras ligam-se à região 3’ não traduzida (3’UTR - untranslated region) - Bloqueia a iniciação da tradução 3) Sítio de ação da regulação gênica regulada por RNA Silenciamento gênico O papel do microRNA – miRNA Produção transitória 60% dos genes humanos chamados como pequenos RNAs temporais (stRNA) Eucariotos superiores (nematódeos, mosca, plantas e mamíferos) Fazem parte de noncoding RNA (ncRNA) incluem snRNA, tRNA, rRNA, snoRNA - Mamíferos codificam mais ncRNA do que mRNA - O miRNA interage com o mRNA na 3’UTR degradação ou inibição da tradução - Controla o desenvolvimento do organismo - Controle de infecções virais em plantas Possuem ~70 nt auto-complementares RNA-RNA grampos duplex Clivagem pela endonuclease DICER ou DROSHA Interação do fragmento com o mRNA alvo em duplex RNA-RNA inibição da tradução ou degradação - Base da técnica de RNA de interferência (siRNA) - Aplicação no controle de infecções virais