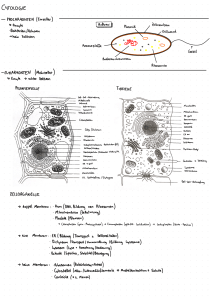
Biologie Klausur Biokatalysatoren: Enzyme beschleunigen Reaktionen , als Enzyme zonin Senkung der Aktivierungsenergie die Energieschwelle, die berschritten werden muss, um Bewegungs-Warme- und Lichtenergie freizusetzen, wird gesenkt. durch die substratspezifisch, kann nur substrate mitkomplementarer molecularer Struktur binden wirkungsspezifisch Katalysieren immer die gleiche Reaktion (gleiches Produkt) - Induced FitModell Das Enzym trittmitdem Substrat in Wechselwirkung und verandert so vorrubergehend die Struktur des aktiven Zentrums Schlssel-Schloss Prinzip Das Enzym istdas Schloss und das Substrat istder Schlussel - substratspezifisch & wirkungsspezifisch Gofaktoren & Coenzyme Cofaktoren:anorganische Metall-lonen (Eisen,Kupfer, Zink & an Enzyme gebunden, for Funktion Coenzyme:komplexe organische Molekule mitdem Substrat an das daher regeneriertwerden lagern sich vorubergehend chemisch und muss notwendig Mangan-lonen) Enzym und verandert sich bei der Reaktion Hydrolyse von ATP an ADP ATP Adenosintriphosphat = Aufban:1 Adeninvest 1 Phosphatkette aus 3 Phosphatgruppen 1 Molekul Ribose ATP: ADP:1 der 3 Phosphatgruppen wird abgespalten um Energie freizusetzen ADPrPhosphor ATP (Abspaltung umkehrbar) Wassermolekule lagern sich aus dem ATP ab durch die Reaktion mitdem Wasser exotherme Reaktion (Warmeenergie) (Hydrolyse) allosterische Effekte (immer reversibel) allosterische Inhibition:Inhibitor bindetsich an das allosterische Zentrum und verandert somitvorubergehend dieStructuresalivenEumsundemmtdieselPerische Zentrum and verander vorubergehee somit die Struktur des aktiven Zentrums und ermglichtdie Umsetzung Hemibel:Inhibitor kann sich kompetetiv vom Enzym ablosen a nichtkompetetiv Inhibitor bindetauerhalb des aktiven Zentrums substrat & Inhibitor konkurrieren erhobte substratkonzentration: Hemmung kann das aktive Zentrum istfrei und deshalb istdie ·berwunden werden Hemmung weniger effektiv erhohte Hemmstoffkonzentration Enzymaktivitt kann uberwunden werden Inhibitor bindetan das aktive Zentrum irreversibel:Inhibitor festam aktiven Zentrum and nichtlosbar, das EnzymverlierSeine Funktion - 11 negative Ruckkopplung Hemmung eines Enzyms durch das Endprodukt der Reaktion, Umsetzung hortaufwenn genugend von Endproduktvorhanden ist Einfluss aufdie Enzymaktivitt Michaelis Menten Konstante (km-Wert) entsprichtder Substratkonzentration,bei der die Halfte der maximalen Reaktionsgeschwindigkeiterreichtist. Substratkonzentration hohere substrationtentration schnellers Reaktionsgeschwindigkeit Geschwindigkeitsteigtab einer bestimmten Substratkonzentration nichtmehr an (alle Enzyme besetzt) 2 3 pH-Wert jedes Enzym hatein bestimmtes pH Optimum auerhalb arbeitetes nur schlechtbzw. gar nichtoder denturiertsogar Temperatur Aktivitat steigtbei Temperaturerhohung (hohere Teilchenbewegung) Hohere Temperaturen konnen zu Instabilitat und somitzur Denturierung fuhren ROT -Regel:18°C verdoppelte Reaktionsgeschwindigkeit Temperaturehohung- Enzymklassen :Oxidurektasen-katalysieren Redoxreaktionen (Elektronentransfer) A B A B- - + - + -Transferasen A B+) - - katalysieren denTransfer funktioneller A+B C > Gruppen (Gruppentransfer) > - ·Hydrolasen Katalysieven hydrolytische Spaltungen (Hydrolyse). - A B+Hc0 > - A-H +B-OH "Lyasen-katalysieven nichthydrolytische Spaltungen (Moleknlspaltung). AB = - AB + ·Isomerasen-katalysieven die Umwandlungin andere A- B-), B-C-A · isomere Strukturen Ligasen-katalysieren Verknipfungen durch ATP-Verbranch (Ligation) A B ATP + Enzyme + in A B +ADPAP = Alltag - Papier, Waschmittel. Leder,Textilien, Pharmazentika (Isomesierung) dem Raumstruktur von Proteinen primustruktur:Aneinanderreihung der Aminusauren in Ketten Sekundrstruktur:Faltung der Polypeptidkette in - Helixund B-Faltblatter (durch Wasserstoffbrckenbindungen zwischen Carboxyl & Aminogruppen stabilisiert) Tertiarstruktur:raumliche Aufban von Proteinen Bindung der Sekundrstrukturen mitSeitenketten Quartiustruktur:mehrere Polypeptidketten lagern sich in einer Untereinheit zusammen Peptidbindung (Elektronenpaarbindungen) Pepfidbindung zwischen bis zu zehn zwei Aminosauven- Dipeptid gebundene Aminosauren, Oligopeptid langere Aminosaurenketten: Polypeptidketten 2 - 18 1007